La Société Française d’Ecologie et d’Evolution (SFE2) vous propose ce Regard de François Bonhomme et Pierre-Alexandre Gagnaire, tous deux directeurs de recherche en génétique à Montpellier, sur le concept d’espèce et la génomique.
.
MERCI DE PARTICIPER à ces regards et débats sur la biodiversité en postant vos commentaires et questions sur les forums de discussion qui suivent les articles; les auteurs vous répondront.
——-
Le concept d’espèce est-il soluble
dans la génomique ?
François Bonhomme (1) et Pierre Alexandre Gagnaire (2)
(1) : Directeur de recherche émérite, Université de Montpellier
(2) : Directeur de recherche, Institut des Sciences de l’Evolution, Montpellier
Regard R128, édité par Anne Teyssèdre
——-
Mots clefs : espèce, concept, génomique, évolution, spéciation, îlots génomiques de divergence
——-
- Résumé
- Espèces en tout genre…
- Pourquoi faut-il essayer de nommer correctement les espèces?
- Le concept dit biologique de l’espèce
- L’apport de la génomique
- Les ilots génomiques de divergence
- Vie et mort des ilots de divergence
- Et l’environnement dans tout ça ?
- Conclusion
- Glossaire
- Remerciements
- Bibliographie
- Regards connexes
- Forum de discussion
—-
Résumé
Depuis au moins deux siècles et pour des raisons tant fondamentales que pratiques, déterminer si deux individus ou deux populations appartiennent à des espèces différentes ou non est l’un des objectifs majeurs de la systématique. Comprendre les mécanismes impliqués dans l’origine des espèces est celui de la biologie évolutionniste depuis ses débuts. Le XXème siècle a vu éclore la génétique de la spéciation, le XXIème est en passe de parachever le travail grâce à la génomique, son avatar moderne. Grâce aux techniques de séquençage de l’ADN à haut débit, il est désormais possible de donner une image précise de la forme que revêt tout au long de leur génome la divergence moléculaire entre deux lignées. Celle-ci est en général tout sauf uniforme, montrant des alternances de zones chromosomiques peu différenciées et d’autres, dites îlots génomique de divergence, où celle-ci est forte. La génomique permet de documenter l’intensité des échanges entre lignées et donc de rendre objectif et mesurable leur degré d’isolement reproductif pour savoir si elles satisfont ou non au concept biologique de l’espèce. Dans la pratique, il existe des cas intermédiaires où les échanges sont absents ou fortement contre-sélectionnés dans les îlots tandis que le reste du génome évolue comme s’il était au sein d’une seule et même espèce. Ces situations, si elles restent difficiles à nommer par un binôme latin, permettent de documenter les mécanismes de spéciation en ouvrant une fenêtre sur l’origine spatiale et temporelle des divergences constatées.
Espèces en tout genre…
La notion d’espèce est vieille comme le monde. Déjà Aristote s’était attaché à vouloir donner une description du monde vivant en hiérarchisant ses catégories à partir de la ressemblance et de la transmission des caractères distinctifs à la descendance. Il est intéressant de noter d’ailleurs que le mot « espèce » s’applique à bien d’autres choses que des taxons du monde biologique. On parle par exemple « d’espèces sonnantes et trébuchantes » pour désigner la monnaie métallique, et le mot « spécifier » ne veut pas dire « créer une nouvelle espèce ». Il existe pour cela le néologisme « spécier » qui a donné « spéciation », lequel lui-même veut aussi bien dire « processus de formation d’une nouvelle espèce biologique » que désigner l’individualisation de formes minéralogiques au cours d’un processus de diagénèse ou de cristallisation. Et ça n’est pas être spécieux que de dire que c’est un mot très spécial ! En fait, ce qui est « spécifique » est ce qui fait la particularité d’un ensemble d’objets, inertes ou animés, et qui en quelque sorte les rassemble ou les identifie par différence avec d’autres ensembles qui n’ont pas les mêmes propriétés. On voit donc d’emblée qu’intuitivement, il s’agit de trouver des discontinuités qui nous permettent d’attribuer un statut distinct et donc « spécifique » pour chaque ensemble ainsi caractérisé.
Pour rester dans le monde des espèces biologiques, ces discontinuités ont été recherchées tout d’abord dans ce qu’il y a de plus immédiat, l’apparence, la forme (la morphologie) et l’expérience commune n’a pas besoin du secours de la taxinomie pour appeler un chat un chat et un chien un chien. Ce qui se distingue sans ambiguïté se nomme sans ambiguïté. L’existence de hiatus clairs et indiscutables dans l’apparence des organismes vivants s’accordait d’ailleurs assez bien avec une vision créationniste et fixiste de l’ordre des choses, les espèces étant vues comme séparées et immuables depuis l’origine des temps. Les choses se sont compliquées quand, après les pionniers du transformisme comme Lamarck et avec les formalisations données plus tard par Darwin, on a reconnu que les espèces ne sont pas immuables et qu’elles ont donc une origine progressive pouvant se traduire sous forme d’arbre de filiation (cités dans Baghdiguian et Bonhomme, 2020).
Cela se complique encore davantage si les discontinuités se font plus subtiles et que l’on cherche à s’appuyer sur d’autres critères que la seule morphologie. Ainsi, le concept d’espèce est un de ceux qui a fait couler le plus d’encre en biologie évolutionniste. On a par exemple parlé de concept typologique, morphologique, écologique, phylogénétique, biologique, génétique…, et on en trouve plusieurs dizaines de définition par exemple dans ce qui peut être considéré comme l’anthologie francophone des définitions de l’espèce (Lherminier et Solignac, 2005).
Pourquoi faut-il essayer de nommer correctement les espèces?
En fait, nous utilisons constamment le terme « espèce » sans toujours savoir ce qu’il recouvre exactement. Or le terme n’est pas neutre et peut influencer notre représentation du monde, de son histoire, voire de son usage. Ainsi par exemple, quand on dit que Neandertal et l’Homme moderne sont deux espèces distinctes, nous n’envisageons pas leurs interactions passées de la même manière que si nous les considérons simplement comme des branches momentanément séparées de notre propre espèce, qui se sont rejointes à des moments de leur histoire. De même, un certain nombre de législations utilisent le rang d’« espèce », selon le système de classification binominal généralisé par Linné en 1758, pour édicter des règles.
Cela pose parfois des problèmes pour déterminer les contours d’une espèce protégée par la loi. Par exemple, si ce que l’on veut protéger n’est qu’une variante régionale (une sous-espèce…) d’un ensemble plus large dans lequel il existe des populations qui ne sont pas menacées, il sera beaucoup plus difficile de lui octroyer un statut de protection reconnu internationalement que si cette entité s’appelle par un nom latin différent du reste.
Cela peut aussi concerner des espèces exploitées, notamment pour édicter des quotas de pêche. Ainsi par exemple, nos travaux passés ont révélé que l’Anchois européen était constitué de deux espèces morphologiquement très semblables et quasi sympatriques* mais présentant de subtiles différences écologiques, l’une d’entre elle étant plus inféodée au milieu côtier et estuarien, l’autre occupant préférentiellement les eaux du large. Or il existe pour les anchois du Golfe de Gascogne des quotas de pêche visant à protéger la ressource de manière indistincte, car un seul nom latin est reconnu par les instances internationales. Ces mesures de gestion des pêches ne sont donc pas adossées à une réalité biologique puisqu’il y a deux entités distinctes, ayant chacune sa propre biologie reproductive et sa propre démographie (Bonhomme et coll., 2022). D’autres exemples similaires abondent, il est donc important de nommer correctement les espèces autant que faire se peut et de savoir quelle réalité biologique cela recouvre.
Le concept dit « biologique » de l’espèce et la génétique
Pour ce qui nous concerne ici, nous nous appuierons tout d’abord sur un exemple très simple qui pose peu de questions, c’est celui de l’interfécondité contrariée entre l’Âne et le Cheval. Bien que nous ne soyons plus à l’heure de la traction hippomobile, tout le monde sait qu’on peut croiser un âne avec une jument, on obtient alors une mule ou un mulet; et dans le sens inverse, un étalon croisé avec une ânesse produira un bardot ou une bardote. Cependant, ces hybrides sont notoirement stériles quoique parfaitement viables. Il n’est donc pas possible de mélanger plus avant les gènes du cheval et ceux de l’âne qui par conséquent appartiennent à deux espèces différentes du fait de cette impossibilité de brassage de leurs patrimoines génétiques respectifs.
Dit dans l’autre sens, selon ce critère, une espèce est un ensemble de populations d’individus pouvant effectivement ou potentiellement se reproduire entre eux et engendrer une descendance viable et féconde, et génétiquement isolé d’autres ensembles similaires. C’est là la base du concept dit biologique de l’espèce (CBE) tel que formalisé par Ernst Mayr au milieu du 20e siècle (Mayr, 1942), mais dont les racines sont bien antérieures, l’interstérilité ayant déjà été considérée comme un critère déterminant par certains auteurs dès le 18eme siècle.
Notons qu’à ce stade, nous n’avons aucunement utilisé les différences morphologiques pourtant évidentes entre l’Âne et le Cheval pour les classer dans deux espèces différentes, leur interstérilité après la première génération nous suffit pour s’assurer de cette impossibilité de remélange. C’est là un exemple extrêmement clair de l’opérationnalité du CBE qu’on pourrait donc qualifier aussi bien de concept génétique, et c’est celui dont nous discuterons les limites tout au long de cet article en le revisitant à la lumière de la génomique.
La biologie moderne a forgé au cours du siècle écoulé des outils qui lui permettent d’aller au-delà de simples descriptions morphologiques pour aborder les bases biologiques des différences interspécifiques. Parmi les disciplines ayant contribué à ce renouveau, la génétique, , occupe une place de choix car elle s’intéresse aux fondements même de ces différences en étudiant la divergence entre populations, au fil des générations, entre populations des gènes ( ou plus exactement des versions de gènes, dites allèles*) dont elles découlentliés à ces différences qui les spécifient. , de leur variabilité (polymorphisme, fréquence des allèles*), leur transmission au fil des générations et leurs variations entre populations (Voir l’encadré introductif sur le génome).
La génétique peut désormais être qualifiée de génomique, car les techniques modernes de séquençage de l’ADN et d’analyse des données permettent maintenant de visualiser ces différences (mutations, polymorphisme, fréquences allèliques*) à l’échelle du génome entier (mutations, polymorphisme, fréquence des allèles*, (voir l’encart introductif ci-dessous sur le génome), c.à.d. au long de chaque chromosome comme cela est illustré par la figure 2 plus bas. Ainsi, la génomique permet de retracer les origines évolutives des différences génétiques et d’en apprécier la transmission d’une génération à l’autre ainsi que leur devenir lors de croisements entre espèces proches (voir plus bas la Figure 3) . Ce faisant, elle permet de cerner les limites mêmes du concept d’espèce, non sans conséquences sur la façon dont nous les désignons…
Encart n°1 : De la cellule au génome
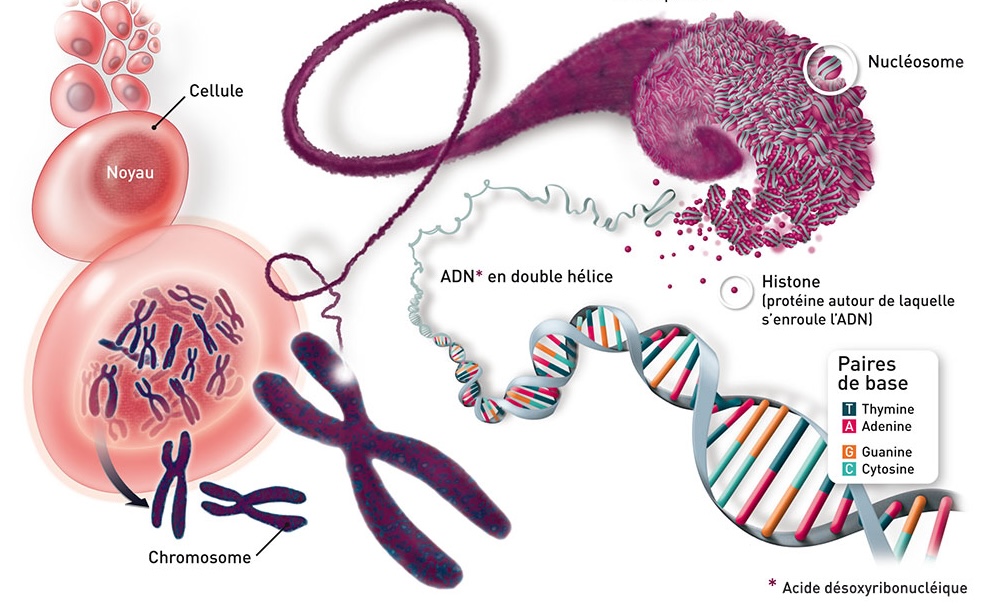
Figure 1 : Représentation schématique du matériel génomique de chaque cellule.
Source : C Les Entreprises du Médicament / M. Phileo et P. Bourcier, 2010.
Chacune de nos cellules corporelle (somatique) renferme un noyau qui contient des chromosomes, n maternels et n paternels, soit 2n chr. = 46 dans le cas de l’espèce humaine. Chaque chromosome contient une longue molécule d’ADN (acide désoxyribonucléique) dite «en hélice double brin», empaquetée dans des protéines et enroulée plusieurs fois sur elle-même. Chaque brin est formé d’une chaîne de nucléotides*, constitués chacun d’un sucre associé à l’une ou l’autre de 4 bases azotées (adénine, thymine, guanine et cytosine), dites A, T, G et C. Ces bases s’associent deux par deux (A avec T, C avec G) pour former des liaisons entre les deux brins de telle sorte que, par exemple, la séquence ATTGTC s’écrira GACAAT sur le brin complémentaire, de polarité inverse.
Dans l’immense majorité des cas, la taille du génome est bien supérieure au strict minimum qui serait nécessaire pour encoder toutes les protéines constitutives d’un organisme. Le génome humain par exemple contient 3 milliards de paires de bases parmi lesquelles environ 1% servent à spécifier les quelques dizaines de milliers protéines de nos cellules. Il peut donc être vu comme une matrice d’ADN dans laquelle sont dispersés les gènes codants pour ces protéines mais également d’autres fonctions liées à sa propre réplication et à la différenciation cellulaire notamment. Pour les protéines, le code génétique contient 64 « codons » constitués de 3 nucléotides* consécutifs chacun.
Quand la molécule est « lue » de la bonne façon par le système de transcription-traduction, les séquences d’ADN sont transcrites en ARN (acide ribonucléique) puis traduites codon par codon en enchaînements d’acides aminés qui les constituent. Le génome contient également des éléments qui n’ont pas nécessairement de fonction connue et qui sont là juste parce qu’ils s’y sont insérés par différents mécanismes. Certains parlent alors d’ADN « poubelle ». Du fait des mutations lors de la réplication, environ 0,25% de ces 3 109 paires de bases différent d’un individu à l’autre, constituant ce qu’on appelle le polymorphisme. La plupart de ces mutations sont dites neutres car situées en dehors des régions codantes du génome et n’ont donc pas de conséquence directe. D’autres peuvent affecter un gène, engendrer des différences entre individus et potentiellement avoir un impact sur leur efficacité reproductive (valeur sélective ou fitness darwinienne), ce qui donne alors prise à la sélection naturelle.
Dans chacune de nos cellules il y a un noyau qui contient des chromosomes, n maternels et n paternels, soit 2n chr. = 46 dans le cas de l’espèce humaine. Chaque chromosome contient une longue molécule d’ADN (acide désoxyribonucléique) dite «en hélice double brin», empaquetée dans des protéines et enroulée plusieurs fois sur elle-même. Chaque brin est formé d’une chaîne de nucléotides*, constitués chacun d’un sucre associé à l’une ou l’autre de 4 bases azotées (adénine, thymine, guanine et cytosine), dites A, T, G et C. Ces bases s’associent deux par deux (A avec T et C avec G) pour former des liaisons entre les deux brins de telle sorte que, par exemple, la séquence ATTGTC s’écrira GACAAT sur le brin complémentaire, de polarité inverse.
Dans l’immense majorité des cas, la taille du génome est bien supérieure au strict minimum qui serait nécessaire pour encoder toutes les protéines constitutives d’un organisme. Le génome humain par exemple contient 3 milliards de paires de bases parmi lesquelles environ 1% servent à spécifier les quelques dizaines de milliers protéines de nos cellules. Il peut donc être vu comme une matrice d’ADN dans laquelle sont dispersés les gènes codants pour ces protéines mais également d’autres fonctions liées à sa propre réplication et à la différenciation cellulaire notamment. Pour les protéines, le code génétique contient 64 « codons » constitués de 3 nucléotides consécutifs chacun.
Quand la molécule est « lue » de la bonne façon par le système de transcription-traduction, les séquences d’ADN sont transcrites en ARN puis traduites codon par codon en enchaînements d’une vingtaine d’acides aminés qui les constituent. Le génome contient également des éléments qui n’ont pas nécessairement de fonction connue et qui sont là juste parce qu’ils s’y sont insérés par différents mécanismes. Certains parlent alors d’ADN « poubelle ». Du fait des mutations lors de la réplication, environ 0,25% de ces 3 109 paires de bases différent d’un individu à l’autre, constituant ce qu’on appelle le polymorphisme. La plupart de ces mutations sont dites neutres car situées en dehors des régions codantes du génome et n’ont donc pas de conséquence directe. D’autres peuvent affecter un gène, engendrer des différences entre individus et potentiellement avoir un impact sur leur efficacité reproductive (valeur sélective ou fitness darwinienne), ce qui donne alors prise à la sélection naturelle.
L’apport de la génomique
Si nous pensons, comme nous le démontre la paléontologie, que l’apparition de nouvelles espèces est un phénomène progressif qui s’inscrit dans le temps long, il y aura nécessairement, entre l’ancêtre commun unique et ses descendants lointains, des situations intermédiaires qui ne seront pas clairement tranchées. Les biologistes sont donc souvent confrontés à des situations beaucoup moins évidentes à appréhender que dans l’exemple de l’Ane et du Cheval précité. C’est le cas par exemple de la Corneille noire et la Corneille mantelée, entre lesquelles il existe une zone de contact qui sillonne l’Europe centrale de l’Italie à l’Ecosse.
Dans cette zone large d’à peine quelques dizaines de kilomètres, un mélange s’opère entre les deux lignées. Côté ouest, les populations présentent le phénotype de la Corneille noire, plus à l’est celui bicolore de la Corneille mantelée, au milieu des phénotypes intermédiaires. La génétique révèle que ces populations intermédiaires constituent des zones d’hybridation dans lesquelles un grand nombre de combinaisons entre les patrimoines génétiques des deux lignées sont présentes (Poelstra et coll., 2014). Chez les corneilles, des gènes (allèles*) sont ainsi échangés entre les deux espèces à la faveur des hybridations répétées qui se produisent le long de la zone de contact.
Ces zones de mélange génétique, dont la largeur peut varier selon les taxons, sont en fait extrêmement fréquentes dans tout le règne animal aussi bien que végétal. Elles résultent le plus souvent de remise en contact de populations ayant été isolées auparavant, par exemple lors d’une période glaciaire ayant fragmenté l’aire de répartition initiale (Hewit, 2000). Ces zones d’hybridation posent donc clairement les limites du concept biologique. A quel endroit exactement mettre le curseur entre une situation claire où de « bonnes » espèces se rencontrent sans s’hybrider (CBE) et des états intermédiaires synonymes d’échanges génétiques plus ou moins intenses ?
C’est là précisément que la génomique permet de rendre objectif l’effet de ces échanges quand ils se produisent (ou non) entre les ensembles considérés. Pour schématiser, l’avancée la plus significative de la dernière décennie a été d’étendre l’analyse des différences génétiques entre espèces d’une poignée de gènes à la totalité du génome, en révélant de surcroît la manière dont ces gènes sont ordonnés le long du génome. En effet, la majeure partie de l’information génétique est organisée de manière linéaire au sein des chromosomes (voir les Figures 1 et 2).
Avec les moyens actuels de séquençage de l’ADN, il est désormais possible d’obtenir la séquence complète des chromosomes d’un individu. On peut ainsi comparer position par position le long de chaque chromosome, les copies homologues d’individus appartenant à des espèces proches. On établit ainsi une carte chromosomique des différences génétiques accumulées au cours du temps entre deux lignées le long du génome. Ce que ce type de carte révèle fréquemment est que la divergence entre espèces proches peut prendre la forme d’un paysage vallonné le long des chromosomes (voir la Figure 2). En effet, il existe des régions fortement différenciées où se concentre la divergence entre lignées, qui contrastent avec un fond de divergence plus faible dans le reste du génome. Ces régions sont couramment nommées « îlots génomiques de divergence ».
Les îlots génomiques de divergence
Afin d’illustrer à quoi peut ressembler concrètement un paysage chromosomique de divergence, considérons celui représenté dans la figure 2, qui concerne la comparaison génomique de deux populations géographiques de poissons étudiées par notre équipe, habituellement regroupées dans la même espèce (Dicentrarchus labrax) appelée Bar dans l’Atlantique et Loup en Méditerranée (Duranton et al, 2018).
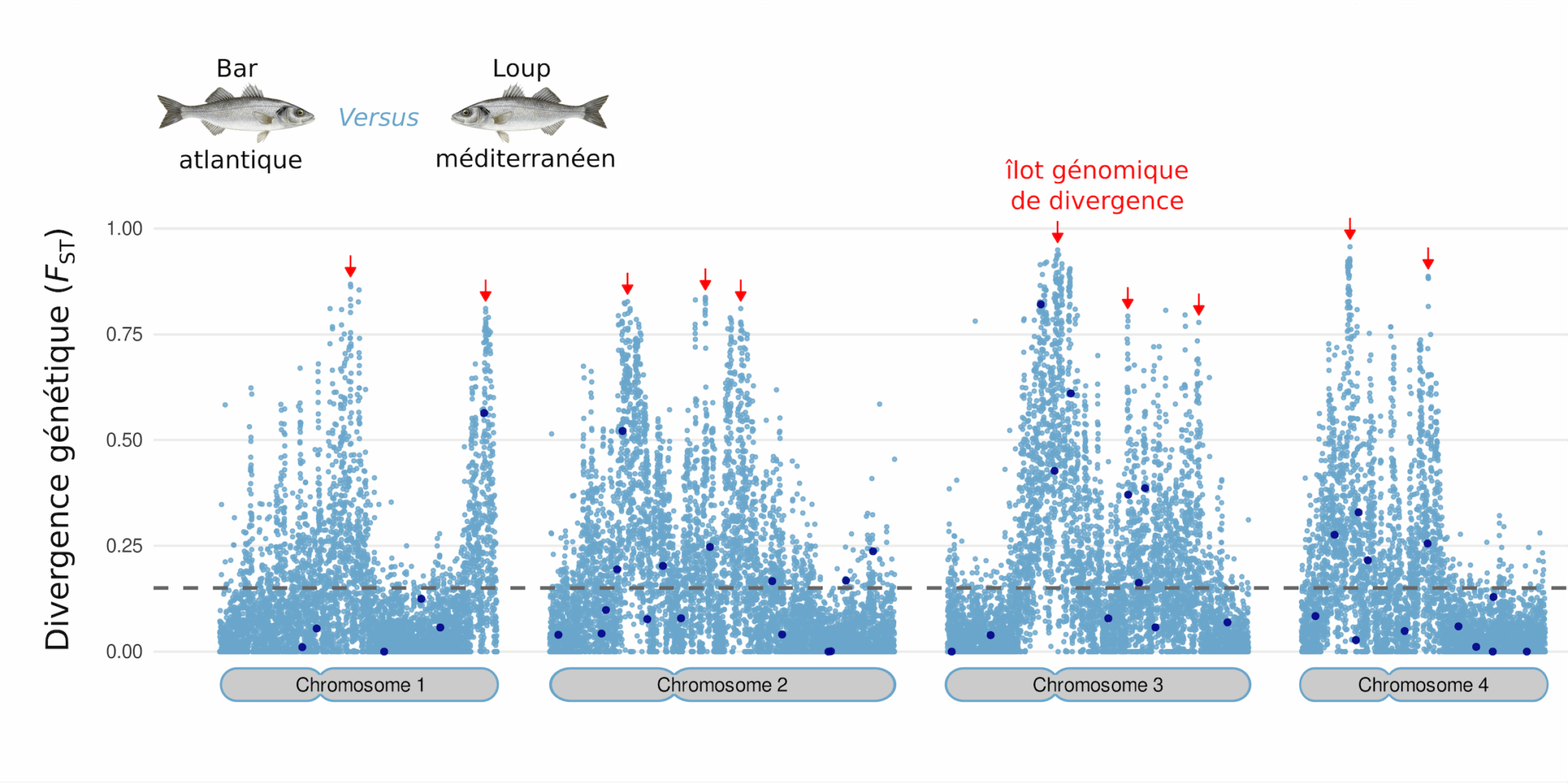
Figure 2 : Mesure de la divergence génétique entre les populations de bars d’Atlantique et de loups de Méditerranée, le long de quatre chromosomes. Source: Duranton et al. 2012.
La mesure employée, le FST, est égale à zéro lorsque les deux populations partagent exactement la même diversité génétique, c’est-à-dire les mêmes variants (allèles*) avec exactement les mêmes fréquences. La valeur maximale de 1 est atteinte lorsque les deux populations ne partagent aucune diversité génétique en commun et présentent donc des variants distincts. Les points bleus représentent la valeur du FST calculée à chaque position variable de l’ADN le long de 4 chromosomes parmi les 24 que compte le génome. La ligne grise pointillée correspond à la moyenne du FST des positions variables du génome. Sur chaque chromosome, les flèches rouges indiquent la présence de régions génomiques où les différences génétiques entre lignées se concentrent. Ces régions sont appelées îlots génomiques de divergence. Les points bleus foncés correspondent à des mesures de divergence obtenues à partir d’une 50aine de positions variables choisie aléatoirement, illustrant notre perception très incomplète de la divergence entre lignées avant l’avènement des techniques de génomique moderne.
La première chose qui saute aux yeux sur cette figure est que la divergence génétique n’est pas distribuée de manière uniforme le long des chromosomes représentés ici, mais qu’il existe des régions beaucoup plus différenciées que les autres (Fig. 2). La présence de valeurs de divergence élevées (proches de FST = 1) dans les régions indiquées par les flèches rouges signifie qu’en ces endroits du génome, les deux lignées ne partagent presque aucun variant génétique (allèle) en commun. Ceci indique qu’elles n’y ont pas échangé d’information génétique depuis un certain temps. En effet, quand deux populations sont complètement isolées, il est impossible qu’elles conservent les mêmes allèles* en même fréquence pendant longtemps, que ce soit par la mutation qui va créer dans l’une et l’autre des variants différents, ou par l’action inexorable de la dérive génétique* ou de la sélection naturelle. Source : Duranton et al. 2012.
À l’inverse, les valeurs de FST proches de 0 dans la plupart des autres régions des chromosomes indiquent qu’à ces endroits du génome, les copies d’ADN sont quasi-identiques dans les deux lignées. De tels endroits semblent indiquer qu’il n’y a pas eu de divergence depuis l’ancêtre commun aux deux lignées, ce qui n’est possible que grâce à des échanges génétiques suffisamment réguliers dans cette partie du génome. Si on ne regardait que les régions où la divergence est proche de zéro, nous serions amenés à conclure que le bar d’Atlantique et le loup de Méditerranée ne forment qu’une seule et même espèce. À l’inverse, si on ne regardait que les îlots de divergence, nous pourrions conclure que les deux entités sont complètement isolées génétiquement depuis longtemps et donc satisfont pleinement aux critères du CBE de Mayr.
Ainsi, les îlots de divergence révélés par la génomique concentrent l’essentiel des différences génétiques entre les lignées. Dans ces régions, parfois également appelées îlots de spéciation, les versions des gènes de l’une et l’autre lignée diffèrent fonctionnellement à cause des mutations distinctes qu’elles ont accumulées au cours du temps. Ceci indique que les deux versions de ces régions génomiques sont partiellement incompatibles et que les individus qui les portent en mélange sont moins aptes à survivre ou à engendrer une descendance que les individus possédant les combinaisons originales de l’une ou l’autre des lignées. Ces génotypes recombinants correspondent aux hybrides produits par croisement entre les deux lignées.
Si ces hybrides étaient stériles ou non viables, cela n’irait évidemment pas plus loin que la première génération, mais c’est très rarement le cas pour les hybrides issus de croisements entre lignées ou espèces proches qui sont généralement viables et, au moins partiellement, fertiles. Quand ces hybrides participent eux-mêmes aux générations successives, un grand nombre de combinaisons génétiques différentes se forment par recombinaison des ascendances (entre séquences d’ADN homologues) au moment de la méiose* (voir la Figure 3 ) et seules certaines d’entre elles passent à travers le crible de la sélection naturelle au fil de générations. Les combinaisons les moins performantes sont éliminées, favorisant ainsi celles qui n’impliquent pas des mélanges d’ascendances dans les îlots de divergence. Les différences génétiques peuvent donc persister voire s’accumuler dans ces îlots, alors qu’elles s’estompent en dehors.
Encart n°2 : Divergence et hybridation entre deux lignées
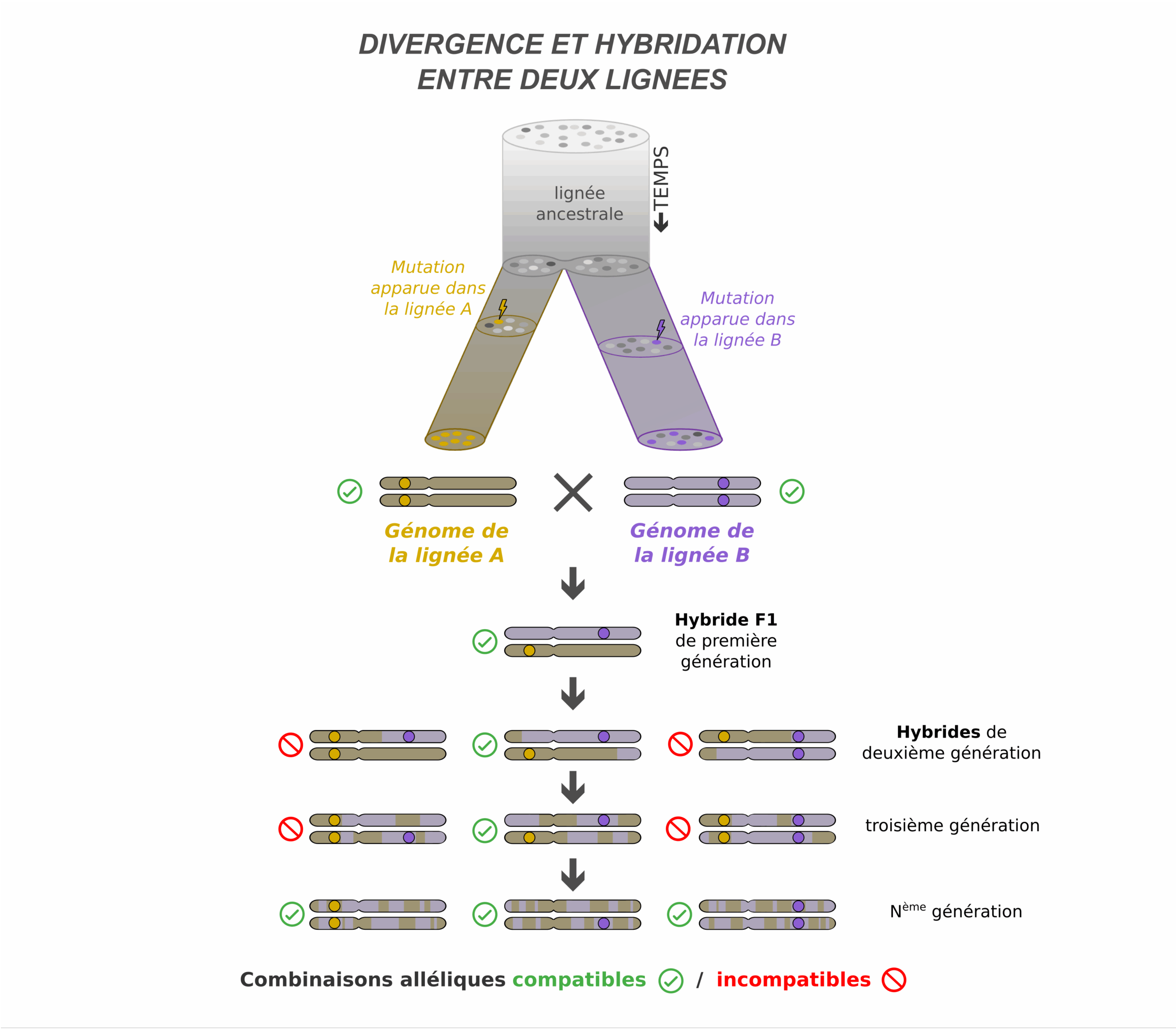
Figure 3 : Recombinaison des ascendances et contre-sélection des combinaisons alléliques* incompatibles. (Auteurs : F. Bonhomme et P-A. Gagnaire, 2025, pour cet article-Regard R128.)
Au cours du processus de divergence, la diversité génétique ancestrale se trie entre les lignées et de nouvelles mutations apparaissent au sein de chaque lignée. Des différences génétiques s’établissent alors entre les génomes des deux lignées, schématisées ici par les mutations jaune et violette. Les croisement hybrides entre lignées génèrent des hybrides de première génération (F1), dont le génome est constitué d’une combinaison d’allèles des deux lignées.
Lors de la gamétogenèse (production des ovules et spermatozoïdes) se produit une division cellulaire particulière appelée méiose*, au cours de laquelle les chromosomes homologues d’origine maternelle et paternelle s’apparient étroitement puis « recombinent » par enjambement (crossing over) de part et d’autre de points de cassure de l’ADN. Les échanges entre les séquences d’ADN homologues se produisent de telle sorte que chaque gamète possède un nouvel assortiment des copies parentales.
Chez les hybrides, la recombinaison a pour effet de mélanger les ascendances des deux lignées, créant au fil des générations une véritable mosaïque d’ascendances recombinées le long des chromosomes. Parmi les nouvelles combinaisons alléliques* produites chez ces hybrides, certaines entraînent une baisse de fertilité ou de survie, qui peut dépendre ou non de l’environnement. Schématiquement, les individus recombinants viables sont ceux qui possèdent au moins une copie de l’allèle ancestral (non coloré sur le schéma ci-dessus) pour chacun des gènes ‘divergents’ considérés, ou qui sont homozygotes* dérivés pour un seul des gènes (i.e. deux copies de l’allèle jaune, ou deux copies de l’allèle violet) et homozygotes ancestraux (deux copies de l’allèle gris) pour l’autre. Au final, les nouvelles combinaisons alléliques* contre-sélectionnées sont celles qui réunissent au sein d’un même génome des mutations apparues dans des lignées indépendantes, mais dont la compatibilité n’a pas été testée au cours l’évolution.
L’homogénéisation génétique causée par la recombinaison érode alors les différences génétiques entre lignées à travers le génome, excepté dans les régions ou la sélection agit en faveur du maintien des combinaisons parentales. Ces régions résistantes aux échanges correspondent aux îlots de divergence de la figure 2.
Cette recombinaison méiotique* permettant le brassage des gènes ne se produit cependant pas avec la même intensité tout au long des chromosomes, où sa fréquence peut varier considérablement entre régions. Certains gènes sont fréquemment échangés et leurs combinaisons brassées dans la population alors que d’autres au contraire sont très liés sur les parties de chromosome qui recombinent très peu. Quand une mutation survient sur un de ces gènes, ceux qui lui sont liés partagent alors un sort commun pour le meilleur comme pour le pire. Si la mutation est avantageuse, le fragment de chromosome pourra voir sa fréquence augmenter avec tous les éléments qu’il contient (même ceux qui sont un peu moins bons que la moyenne, c’est la « note globale » qui compte).
Ces zones de faible recombinaison sont donc aussi des zones d’accumulation rapide de divergence fonctionnelle, parce que les gènes qu’ils contiennent agissent en synergie et que leur association « résiste » mieux à la force homogénéisatrice de la recombinaison chromosomique. Les mécanismes méiotiques ne sont cependant pas infaillibles et il arrive parfois qu’un bout de segment chromosomique soit réinséré à l’envers dans le chromosome après un double événement de recombinaison. Ce fragment inversé porte exactement les mêmes gènes que son homologue non-inversé, mais il ne peut plus s’apparier correctement avec lui au moment de la méiose, ce qui bloque encore plus la recombinaison à cet endroit du génome.
Quand cette méiose* est défectueuse, elle peut en outre conduire à des formes (gamétiques) aberrantes induisant des stérilités complètes ou partielles qui sont souvent à la base de l’isolement reproductif entre espèces. Dans les faits, les îlots de divergence coïncident presque toujours avec des zones de faible recombinaison. Parce que les gènes qu’ils contiennent affectent en synergie la survie et la fécondité des hybrides, ce sont aussi des zones d’accumulation rapide de la divergence entre lignées. Il en va fréquemment ainsi pour les chromosomes sexuels, qui généralement recombinent peu. De plus, selon la règle de Haldane (1922), ces stérilités touchent préférentiellement le sexe dit hétérogamétique quand il y en a un (mâles XY chez les mammifères, femelles ZW chez les oiseaux…).
Certaines espèces emblématiques (Epinoches, Gobe-mouches, Papillons du genre Heliconius, Cichlidés…) ont fourni voici une douzaine d’années les premiers exemples d’architecture génomique montrant l’existence d’îlots de divergence. (ex: Ellegren et al 2012, Hohenlohe et al 2012, Nadeau et al 2012, Malinsky et al 2015). La très grande majorité des études produites depuis révèle la présence de tels îlots – le mécanisme décrit ci-dessus est donc très général -, néanmoins leur étendue chromosomique et leur nombre est très variable d’un cas à l’autre. Sans surprise, les comparaisons entre lignées ou espèces qui nous apparaissent les plus distantes selon des critères reproductifs, morphologiques ou adaptatifs sont aussi celles qui divergent le plus au niveau de leur séquence d’ADN. Les études génétiques nous éclairent sur l’étendue génomique de cette divergence et sur celle des régions qui semblent toujours être librement échangées entre espèces. Mais elles donnent une image de la divergence génétique au temps t, sans en expliquer l’origine ni les mécanismes moléculaires associés.
Vie et mort des îlots de divergence
L’identification des îlots génomiques de divergence résulte par définition de la comparaison de deux lignées évolutives divergentes. La cause de cette divergence la plus facile à comprendre est l’isolement géographique, ou allopatrie*. Les nombreuses fluctuations climatiques du Quaternaire ont entraîné pour un très grand nombre de lignées des cycles de contraction /expansion de leurs aires de répartition, aussi bien sous les tropiques que dans les zones tempérées. Quand l’isolement géographique interrompt les échanges génétiques au cours d’une phase de contraction, la divergence moléculaire est inévitable comme cela a été évoqué plus haut. Si l’isolement géographique est rompu au cours d’une phase de ré-expansion, les gènes des deux lignées se retrouvent co-exprimés chez les individus hybrides. Dès lors, deux issues sont possibles:
Soit la divergence fonctionnelle des gènes est faible et les hybrides de premières générations sont suffisamment fertiles et produisent des descendants eux-mêmes fertiles et portant des génomes recombinés. On peut alors assister à un brassage complet des pools génétiques des deux lignées, les combinaisons les plus avantageuses prenant le dessus; soit la divergence est plus forte et, bien que les hybrides de premières générations soient encore plus ou moins fertiles, cela révèle des incompatibilités liées à certaines portions du génome. Ces incompatibilités, dues la plupart du temps à des perturbations de l’expression correcte des gènes ou de leur bon fonctionnement au sein des réseaux moléculaires, rendent les individus porteurs de nouvelles combinaisons alléliques* moins performants (Figure 3). Certaines combinaisons alléliques non existantes dans les lignées d’origine seront donc systématiquement écartées par la sélection naturelle et le remélange complet des génomes ne pourra alors pas s’effectuer.
Il va alors se former lors du contact entre ces populations divergentes une zone d’hybridation où seules les régions du génome qui ne contiennent pas de telles incompatibilités peuvent diffuser de part et d’autre de cette zone géographique après recombinaison. Par contraste, les zones du génome qui ne diffusent pas dans les populations adjacentes et qui restent bloquées dans la zone d’hybridation constituent ce qu’il est convenu d’appeler des îlots de divergence. Ces îlots, en nombre et étendue variable selon l’intensité de la divergence initiale, sont donc pratiquement étanches aux échanges d’ADN.
NB : Notons toutefois que si un îlot d’une lignée était toujours « meilleur » à l’état homozygote que celui de la lignée à laquelle il se trouve confronté lors d’un contact secondaire, alors le bloc entier pourrait théoriquement envahir cette autre lignée de telle sorte que la trace en soit totalement perdue, puisqu’il n’y aurait plus de différenciation à cet endroit du génome. Certains exemples peuvent être trouvés, notamment pour des segments d’ADN contenant des gènes conférant une résistance accrue à un pathogène ou un parasite.
Selon ce scénario d’isolement géographique, le moteur de la création de ces îlots est ainsi la divergence qui s’accumule entre les différentes lignées quand elles se retrouvent isolées par la contraction des aires initiales de répartition. Au prochain cycle de contraction/expansion, de nouvelles divergences moléculaires pourront se produire et venir s’ajouter aux précédentes, ce qui renforcera encore les incompatibilités génétiques lors de la remise en contact, jusqu’à ce qu’elles deviennent si fortes qu’on obtienne un isolement reproductif complet. Ce mécanisme de spéciation par allopatrie* temporaire ou récurrente – renforcée par la moindre valeur sélective des hybrides dans les zones de contact – rejoint donc celui de l’allopatrie prolongée, pour éventuellement aboutir à deux bonnes espèces satisfaisant sans discussion possible au critère mayrien du concept biologique.
Et l’environnement dans tout ça ?
Au-delà des mécanismes de la méiose et de la recombinaison génétique, ce qui précède n’a fait appel qu’à la notion d’isolement géographique et à l’incontournable divergence moléculaire qui s’ensuit, comme si les deux lignées avaient été confrontées à des environnements identiques et subissaient exactement les mêmes pressions écologiques dans leurs habitats respectifs lors des phases d’allopatrie. Bien entendu, ceci ne se produit jamais dans la réalité et les lignées subissent des trajectoires adaptatives différentes. Quand elles sont remises en contact, elles n’ont jamais exactement les mêmes préférendums écologiques.
Si les hybrides et les recombinants sont clairement moins performants car éloignés de l’optimum adaptatif de l’une ou l’autre lignée, ceci permet le maintien, voire le renforcement de la spécialisation écologique de chaque lignée permettant la coexistence de ce qui est parfois appelé des écotypes. Les écotypes sont en quelque sorte des versions locales de lignées divergentes adaptées à des environnements contrastés auxquels elles sont préférentiellement inféodées. Cela peut-être des écotypes différant par exemple entre un environnement lacustre et une rivière, entre plaine et montagne, ou bien encore entre deux plantes hôtes, etc.
Néanmoins, reconnaître l’existence d’écotypes n’implique pas a priori de connaître leur niveau d’isolement reproductif. S’il est faible, ces écotypes peuvent ne pas perdurer dans le temps et se remélanger ou disparaître à la première perturbation environnementale. Un exemple célèbre est fourni par certains poissons cichlidés très colorés du lac africain Victoria, où il existe des morphes de couleurs différentes qui s’accouplent préférentiellement (phénomène dit d’homogamie*) et se partagent le milieu sur des différences subtiles de profondeur et d’alimentation. Lorsque le lac s’est eutrophisé* suite à l’introduction d’un prédateur, les eaux sont devenues troubles à cause d’une croissance planctonique décuplée, et certains de ces morphes colorés n’ont pas pu maintenir visuellement leur isolement comportemental, occasionnant une dédifférenciation de ces écotypes (Seehausen et al. 1997).
Ce n’est donc pas parce qu’on identifie l’existence d’écotypes que ceux-ci sont irrémédiablement engagés sur la voie d’une spéciation complète, et leur attribuer un statut d’espèces à part entière méritant une désignation latine binominale peut porter à conséquences (réglementaires notamment).
À l’inverse, dans le cas des anchois mentionnés au début de cet article, la stabilité des deux écotypes est attestée par l’existence de la paire « anchois côtiers / anchois hauturiers » dans de très nombreuses répliques géographiques, depuis la mer Noire jusqu’à la mer du Nord. Ceci malgré l’existence de nombreux hybrides et le partage d’une fraction importante de leur génome en dehors des îlots de divergence. Quoiqu’en violation apparente du concept biologique, on peut donc considérer que ces écotypes ont atteint fonctionnellement le stade d’espèces puisqu’ils perdurent en quasi-sympatrie jusqu’à preuve du contraire. Il n’est cependant pas possible de savoir dans quel sens leur niveau d’isolement reproductif pourrait évoluer lors du prochain cycle glaciaire…
Conclusion
Comme on vient de le voir, la génomique permet dans de nombreuses situations qu’on pourrait qualifier d’intermédiaires entre une seule espèce et deux « bonnes espèces » manifestement interstériles (comme l’Âne et le Cheval), de rendre objectif et mesurable le degré d’isolement reproductif entre deux populations. Elle nous dit également que cette divergence n’affecte pas de manière homogène le patrimoine génétique des populations et des individus, et qu’il existe des grandes différences entre ce qu’il est convenu d’appeler des îlots génomiques de divergence pour lesquels tout se passe comme si l’isolement génétique entre les lignées était total, et le reste du génome qui évolue comme s’il était au sein d’une seule et même espèce.
Il n’y a pas de terminologie stabilisée pour ce genre de situation pourtant très fréquente (on parle parfois dans la littérature de semi-espèces ou de quasi-espèces). Il n’est donc pas choquant dans ce cas-là de les désigner par un binôme latin, faute de mieux, mais nous touchons-là du doigt les limites du CBE. La génomique permet en outre de documenter les mécanismes de spéciation en ouvrant une fenêtre sur l’origine spatiale et temporelle des divergences constatées.

Figure 4 : Gobe-mouche noir (Cliché Ron Knight, CC BY 2.0).
La comparaison génomique de deux espèces de gobe-mouches aux répartitions marginalement chevauchantes, le Gobe-mouche noir Ficedula hypoleuca (au Nord et à l’Ouest de l’Europe) et le Gobe-mouche à collier F. albicollis (Europe Est et Sud), a mis en évidence une cinquantaine de pics ou îlots de divergence (Fst > 0,7) sur un fond de moindre divergence (Fst < 0,4), soutenant l’hypothèse d’une alternance de phases d’isolement géographique et donc reproducteur (en allopatrie) pendant les glaciations du Quaternaire, et de phases d’hybridation lors des périodes de sympatrie interglaciaires (AT, cf. Ellegren et al, 2012).
Glossaire
- Allèlique, alléle : Deux versions différentes d’un même gène, donc situées au même endroit sur le chromosome, sont dites alléliques. Un allèle est un variant allélique, il peut être caractérisé par sa fréquence dans la population.
- Allopatrie : Fait d’être situé dans des aires géographiques distinctes et non-contigües, par opposition à sympatrie. Les espèces allopatriques n’ont donc pas de contact direct
- Dérive génétique : Caractérise les changements de fréquence liés au tirage aléatoire des variants alléliques à chaque génération. C’est donc un processus totalement neutre et non directionnel par opposition à la sélection naturelle. Plus le nombre d’individus reproducteurs est faible, plus la dérive est rapide, pouvant aboutir à l’élimination de certains allèles et à la fixation d’autres.
- Eutrophisation : Augmentation de la capacité trophique d’un milieu aquatique résultant d’un enrichissement important en nutriments tels que nitrate et phosphate. Il en résulte une croissance bactérienne, algale et planctonique accrue, augmentant la turbidité du milieu.
- Eucaryotes : ensemble des organismes (uni- ou pluricellulaires) aux cellules dotées d’un compartiment -dit noyau- renfermant un ou deux lots de chromosomes, selon le type de cellule (sexuelle ou somatique) : animaux, végétaux, champignons, protistes, algues.. (AT)
- Eutrophisation : Augmentation de la capacité trophique d’un milieu aquatique résultant d’un enrichissement important en nutriments tels que nitrate et phosphate. Il en résulte une croissance algale et planctonique accrue augmentant la turbidité du milieu.
- Gamète : cellule sexuelle reproductrice -ovule ou spermatozoïde- des eucaryotes, dotée d’un seul lot de chromosomes (i.e. « haploïde ») et non pas de deux lots comme leurs autres cellules (dites diploïdes, issues de la fusion de deux gamètes parentaux (fécondation d’un ovule par un spermatozoïde). (AT)
- Hétérozygotie : possession de deux versions différentes (allèles*) d’un même gène, héritées des deux parents, par les cellules à deux lots de chromosomes (dites diploïdes) d’un organisme eucaryote* (animal, végétal, protiste, …). (AT)
- Homogamie : quand la reproduction sexuée se fait préférentiellement entre partenaires partageant les mêmes caractéristiques phénotypiques. (Antonyme : hétérogamie.)
- Homozygotie : possession de deux versions identiques (i.e. même allèle*) d’un gène, héritées des deux parents, par les cellules à 2n chromosomes d’un organisme eucaryote*. (AT)
- Méiose : La méiose est le mode de division cellulaire des eucaryotes* qui conduit à la formation des cellules sexuelles, ou gamètes. Elle transforme une cellule diploïde (dotée de 2n chromosomes) en 4 cellules haploïdes (n chromosomes). La méiose débute par une phase d’appariement au cours de laquelle les chromosomes homologues (d’origine paternelle et maternelle) recombinent et forment ainsi des associations génétiques nouvelles.
- Nucléotide : Elément constitutif des acides nucléiques. Le sucre associé à un groupement phosphate et aux 4 paires de bases azotées est le désoxyribose (pour l’ADN) ou le ribose (pour l’ARN).
- Parapatrie : Situation intermédiaire entre sympatrie et allopatrie, quand les aires géographiques de deux espèces sont distinctes mais adjacentes et que des contacts restent possibles
- Sympatrie : Fait d’être situé dans des aires géographiques chevauchantes, par opposition à allopatrie. Les individus des espèces sympatriques peuvent donc être en contact direct.
Remerciements:
Nous tenons à remercier chaleureusement les membres de l’équipe avec lesquels nous avons vécu l’aventure passionnante de la génomique évolutionniste depuis ses balbutiements, ainsi qu’Anne Teyssèdre pour sa relecture avisée du manuscrit et son travail d’édition.
Bibliographie
- Baghdiguian S. & Bonhomme F., 2020. Arborescences, aux origines de l’arbre de vie. Espèces 35, 50-57.
- Bonhomme F., Meyer L., Arbiol C., Bănaru D., Bahri-Sfar L., Fadhlaoui-Zid K., Strelkov P., Arculeo M., Soulier L., Quignard J.-P. & Gagnaire, P.-A., 2022. Systematics of European coastal anchovies (genus Engraulis Cuvier). Journal of Fish Biology 100.
- Duranton M., Allal F., Fraïsse,C., Bierne N., Bonhomme F. & Gagnaire P. A., 2018. The origin and remolding of genomic islands of differentiation in the European sea bass. Nature communications 9(1), 2518.
- Ellegren H., Smeds L., Burri R. et al., 2012. The genomic landscape of species divergence in Ficedula flycatchers. Nature 491(7426), 756-760.
- Haldane J.B.S., 1922. Sex ratio and unisexual sterility in hybrid animals, J. Genet. 12 (2), 101–109.
- Hewitt G., 2000. The genetic legacy of the Quaternary ice ages. Nature 405 (6789), 907-913.
- Hohenlohe P. A., Bassham S., Currey M., & Cresko W. A., 2012. Extensive linkage disequilibrium and parallel adaptive divergence across threespine stickleback genomes. Philosophical Transactions of the Royal Society B: Biological Sciences,367(1587), 395-408.
- Lherminier P, Solignac M., 2005. De l’Espèce, Editions Syllepse (Paris) 681pp.
Nadeau N.J., Whibley, A., Jones, R. T., Davey et al., 2012. Genomic islands of divergence in hybridizing Heliconius butterflies identified by large-scale targeted sequencing. Phil. Trans. R. Soc. B 367, 343–353. - Malinsky, M., Challis, R. J., Tyers, A. M., Schiffels, S., Terai, Y., Ngatunga, B. P., … & Turner, G. F. (2015). Genomic islands of speciation separate cichlid ecomorphs in an East African crater lake. Science 350(6267), 1493-1498.
- Poelstra, J. W., Vijay, N., Bossu, C. M., Lantz, H., Ryll, B., Müller, I., … & Wolf, J. B. (2014). The genomic landscape underlying phenotypic integrity in the face of gene flow in crows. Science 344(6190), 1410-1414.
- Seehausen O., Alphen J.J.V. & Witte F., 1997. Cichlid fish diversity threatened by eutrophication that curbs sexual selection. Science 277(5333), 1808-1811.
Regards connexes :
- Regards sur l’évolution : https://sfecologie.org/tag/evolution/
- Regards sur l’adaptation : https://sfecologie.org/tag/adaptation/
- Regards sur les mécanismes : https://sfecologie.org/tag/mecanismes/
- Regards sur méthodes et outils : https://sfecologie.org/tag/methodes-et-outils/
—–
Article édité et mis en ligne par Anne Teyssèdre.

Commentaires récents