Résumé
Les activités humaines entraînent un appauvrissement global de la biodiversité, en particulier chez les vertébrés terrestres. Dans ce contexte, il devient essentiel de développer des méthodes de biosurveillance efficaces, capables d’évaluer l’impact des actions de conservation et de restauration écologique. La forêt des Landes, dominée par une monoculture de pins maritimes, constitue un cadre pertinent pour tester ces approches, d’autant que des plantations de haies de feuillus y sont envisagées pour diversifier les essences, bien que les effets écologiques de ces aménagements restent à documenter.
Une approche prometteuse de détection de la présence d’animaux repose sur l’analyse de l’ADN environnemental (ADNe), c’est-à-dire l’ADN que les organismes excrètent naturellement dans leur environnement. Récemment, les toiles d’araignées ont été explorées comme un substrat original pour collecter cet ADNe. Véritables pièges adhésifs naturels, elles sont capables de capter des particules en suspension dans l’air, pouvant contenir des traces d’ADN d’organismes passés à proximité. Leur utilisation pour détecter des vertébrés est récente mais montre un fort potentiel pour la biosurveillance. Plusieurs questions restent néanmoins ouvertes : comment les caractéristiques des toiles (forme, hauteur…) influencent-elles la détection ? Quelle est la complémentarité avec les méthodes traditionnelles ? Et les toiles permettent-elles de détecter des variations fines de biodiversité en lien avec le paysage ? Dans cette étude, des toiles ont été collectées sur 51 sites de la forêt des Landes répartis entre trois types d’habitats (lisières de pins, haies de feuillus et forêts de feuillus). Chaque toile a été prélevée à différentes hauteurs, sa forme notée, puis des outils génétiques ont été utilisés pour tenter de détecter à la fois l’ADN des vertébrés et celui des araignées à l’origine des toiles.
Des analyses préliminaires, menées sur 156 toiles, ont permis de détecter l’ADN de nombreuses espèces de vertébrés sauvages : majoritairement des oiseaux, mais aussi des mammifères, reptiles et amphibiens. La richesse en espèces détectées était plus élevée en forêt de feuillus, suivie des haies, puis des lisières de pin, ce qui reflète bien le gradient attendu de restauration écologique. L’identification génétique des araignées a été plus limitée, avec seulement 18 % des échantillons associés à une espèce d’araignée, vraisemblablement en raison d’un manque de spécificité du marqueur génétique employé. Toutefois, l’approche basée sur l’observation des formes de toiles reste prometteuse pour élucider les conditions favorables à la détection d’ADNe de vertébrés.
Ces premiers résultats confirment que les toiles d’araignées permettent la détection d’un large spectre de vertébrés et reflètent des différences de biodiversité en lien avec les types d’habitats. Ainsi, elles apparaissent comme un outil complémentaire aux méthodes classiques, avec un fort potentiel pour les suivis multi-taxonomiques non invasifs. Les analyses en cours à partir des échantillons collectés dans le cadre de cette bourse permettront de préciser les facteurs influençant la détection et de mieux évaluer leur place dans les stratégies de biosurveillance à venir.
Justifications
Les activités humaines ont provoqué un déclin généralisé des vertébrés terrestres et de nombreuses extinctions (1). Parmi les pressions majeures, les transformations du paysage et la conversion des habitats constituent le principal moteur de la perte de biodiversité mondiale (2). Face à cette situation, la mise en place de systèmes de biosurveillance efficaces est plus que jamais nécessaire pour prévenir la dégradation des écosystèmes, orienter les efforts de conservation et guider les initiatives de restauration.
La forêt des Landes est l’un des plus grands massifs forestiers entièrement façonnés par l’humain. Composée presque exclusivement de pins maritimes (Pinus pinaster) plantés à des fins industrielles, elle est un cas extrême de sylviculture intensive à la biodiversité appauvrie. Dans ce contexte, la diversification des essences d’arbres apparaît comme une stratégie prometteuse pour recréer des habitats favorables à la biodiversité. Parmi les solutions envisagées, la plantation de haies de feuillus se présente comme une approche de restauration permettant d’enrichir les écosystèmes tout en minimisant l’impact sur les activités économiques. Toutefois, l’impact réel de ces structures paysagères sur la biodiversité reste à quantifier. Ma thèse s’inscrit dans cette problématique, en se focalisant sur les communautés de petits mammifères et leur suivi par piégeage. Cependant, l’influence des modifications paysagères ne se limite pas à un seul groupe faunistique. La mise au point de méthodes de suivi multi-taxonomiques efficaces et complémentaires permettrait de grandement améliorer les capacités de biosurveillance et d’évaluer l’impact de cette stratégie de restauration.
Ces dernières années, le séquençage de l’ADN par metabarcoding d’échantillons environnementaux a émergé comme une méthode prometteuse pour surveiller la biodiversité. Contrairement aux méthodes traditionnelles de suivi, qui peuvent être logistiquement complexes et limitées à certaines espèces, cette approche permet la détection simultanée d’un large éventail de taxons à partir de traces d’ADN (3). L’ADN environnemental (ADNe) utilisé dans les méthodes de suivi correspond généralement à l’ADN total extrait d’un type d’échantillon spécifique appelé « substrat » (4,5). Ce dernier joue un rôle clé, car il influence la probabilité de détection et les composantes de biodiversité détectables (6). En effet, le substrat détermine les propriétés physico-chimiques dans lesquelles l’ADN se maintient, ce qui impacte sa vitesse de dégradation (7). En outre, certaines espèces interagissent plus fréquemment avec certains substrats que d’autres en raison de leur comportement et de leur écologie (8).
La détection d’espèces à partir de l’ADNe présent dans l’air ouvre récemment de nouvelles perspectives pour la surveillance de la biodiversité terrestre. Grâce à l’ubiquité de l’air et à la dispersion naturelle des particules en suspension, l’analyse de substrats capable de retenir l’ADNe de l’air pourrait permettre des évaluations larges, non ciblées et non invasives de la biodiversité (9). L’ADNe aéroporté peut être collecté à l’aide de méthodes actives ou passives. L’échantillonnage actif consiste à aspirer artificiellement l’air à travers un filtre à l’aide d’un dispositif motorisé, tandis que l’échantillonnage passif repose sur le dépôt naturel de particules sur une surface. Les méthodes passives de collecte d’ADNe aéroporté pourraient offrir plusieurs avantages potentiels. Bon nombre évitent notamment le recours à des installations, permettant de collecter plusieurs échantillons en une seule session. Elles pourraient ainsi s’avérer particulièrement adaptées aux évaluations rapides de biodiversité ou à l’étude de sites difficiles d’accès. De plus, l’accessibilité des substrats capables de capter passivement l’ADNe aéroporté ouvrir la voie à leur intégration dans des programmes de science citoyenne, élargissant ainsi les capacités de surveillance à large échelle.
Dans ce contexte, les toiles d’araignées émergent comme un substrat prometteur pour la collecte d’ADNe aéroporté. Faciles à prélever et présentes dans divers milieux naturels ou anthropisés, ces structures ont été sélectionnées pour servir de pièges adhésifs (10). Des travaux récents ont montré qu’elles pouvaient capturer efficacement l’ADNe de vertébrés (11,12). De plus, nous avons montré que les toiles offrent, dans certains contextes, une probabilité de détection supérieure à celle d’autres substrats comme le sol ou la surface de feuilles, renforçant ainsi leur potentiel pour la surveillance des vertébrés terrestres (12).
Plusieurs aspects clés restent à explorer pour optimiser l’usage des toiles d’araignées dans la biosurveillance par ADNe. i) Un premier enjeu concerne l’influence des caractéristiques des toiles sur la détection des espèces. Les toiles varient fortement en forme, composition et emplacement selon les espèces d’araignées, ce qui pourrait affecter la quantité et le type d’ADNe accumulé. Comprendre ces effets est essentiel pour identifier les critères à standardiser et améliorer la reproductibilité des études futures. ii) Un second point concerne l’évaluation de la complémentarité et de la valeur ajoutée de cette approche par rapport aux méthodes traditionnelles de suivi, afin d’identifier les contextes dans lesquels elle pourrait être intégrée de manière pertinente aux dispositifs de surveillance existants. iii) Enfin, alors que la biosurveillance par ADNe en milieu terrestre s’est jusqu’ici concentrée sur des échelles spatiales larges pour dresser des inventaires généraux, les toiles d’araignées pourraient permettre de détecter des variations locales de biodiversité associées à la structure et à la composition du paysage. Cette capacité reste à démontrer et comparer aux résultats fournis par les méthodes traditionnelles.
Pour répondre à ces problématiques nous avons cherché à i) identifier si certains types et hauteurs de toiles offrent une meilleure probabilité de détection d’ADNe de vertébrés à partir d’une approche typologique et de données moléculaires sur les familles d’araignées à l’origine des toiles. ii) Comparer les communautés de vertébrés détectées à partir des toiles avec celles observées par les méthodes traditionnelles afin d’évaluer la complémentarité et la valeur ajoutée de cette approche en biosurveillance. iii) Explorer le potentiel des toiles d’araignées pour révéler des variations de biodiversité en lien avec les types d’habitats et les gradients paysagers, notamment dans le contexte de la restauration écologique de la forêt des Landes.
Méthodes
Échantillonnage
L’échantillonnage a été réalisé dans une zone de 1 050 km² de la forêt des Landes, sur 51 sites englobant trois types d’habitats : les lisières de pins (12 sites), les haies de feuillus (23 sites) et les forêts de feuillus (16 sites). Les lisières et les haies sont également subdivisées en 6 catégories paysagères en fonction de leur connectivité et du pourcentage de feuillus environnant (Figure 1). Un site témoin pour lequel la faune vertébrée captive est connue a également été inclus (parc zoologique de Montpellier) afin de vérifier la capacité de nos protocoles à détecter les espèces présentes localement (contrôle positif).
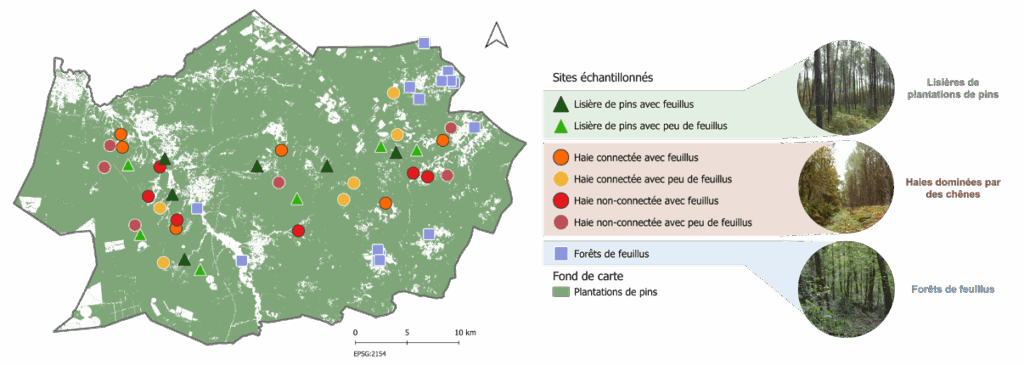
Figure 1 : Carte des sites d’échantillonnage dans la forêt des Landes (France). Le couvert en conifères est une production du CNES pour le centre de données Theia (www.theia-land.fr) à l’aide des produits Copernicus.
Les toiles d’araignées ont été collectées individuellement sur chaque site à l’aide d’anses d’inoculation en plastique stérile, puis transférées dans des tubes de 2 ml. Trois toiles ont été prélevées par site à trois hauteurs différentes (cheville, genou et torse). Pour chaque toile, la hauteur de prélèvement a été notée, ainsi que le type d’architecture observé (entonnoir, nappe, orbiculaire, réseau ou autre) et une estimation semi-quantitative de la quantité de matière prélevée (faible, moyenne ou grande). Les toiles ont été conservées à sec et stockées à -20 °C dans un délai de moins de huit heures après leur collecte.
Biologie moléculaire
Pour éviter toute contamination, toutes les extractions ont été réalisées dans un laboratoire exempt d’ADN de vertébrés extrait ou amplifié. Les toiles d’araignées ont été extraites à l’aide du kit DNeasy Blood & Tissue (Qiagen) conformément aux recommandations de Newton et al. (2025) (13).
L’amplification par PCR de l’ADNe mitochondrial de vertébrés a été réalisée à l’aide des amorces universelles 12S-V5-F et 12S-V5-R ciblant un fragment de 100 pb du gène 12S rRNA, ainsi que 16Smam1-F et 16Smam2-R ciblant un fragment de 96 pb du gène 16S. L’amplification de l’ADNe des araignées a été réalisée à l’aide des amorces MG2-LCO1490 et MG2-univ-R ciblant un fragment de 133 pb du gène COI.
Chaque échantillon d’ADN a été analysé trois fois indépendants pour chaque gène, avec l’ajout de contrôles négatifs d’extraction, de PCR et d’indexation pour respectivement augmenter la sensibilité de détection et s’assurer de l’absence de faux positifs. Les bibliothèques de séquençage ont été préparées par une méthode de PCR en deux étapes, comme décrit dans Galan et al. (2018), et en utilisant des index doubles uniques (UDI) pour pouvoir séquencer les échantillons ensembles tout en assurant une forte traçabilité de l’appartenance de chaque séquence à un échantillon. Le séquençage a été réalisé en paire de 2 x 150 pb sur une plateforme NextSeq (Illumina).
Bio-informatique et statistiques
Les séquences ont été analysées à l’aide du pipeline FROGS (Find, Rapidly, OTUs with Galaxy Solution) (14) afin de regrouper les séquences sous forme de variants de séquence d’amplicons (ASV). Après nettoyage des données, les ASV restants ont été comparés à la base de données GenBank pour attributions taxonomiques à l’aide de l’outil BLASTN. Des pourcentages d’identité supérieurs à 98 % ont été jugés suffisants pour l’attribution au niveau de l’espèce lorsqu’une seule espèce présentait la meilleure correspondance.
Le dernier lot d’échantillons étant en cours de séquençage, les résultats ci-dessous sont préliminaires et partiels. Les analyses à venir s’appuieront sur des régressions logistiques à effets mixtes pour modéliser la probabilité de détecter un vertébré en fonction des facteurs étudiés (hauteur, typologie, quantité et catégorie d’habitats). La comparaison avec les méthodes traditionnelles sera effectuée en utilisant les données de capture de petits mammifères issues de ma thèse (projet BePrep : https://www.beprep-project.eu/), ainsi que les suivies ornithologiques et herpétologiques réalisés dans le cadre du projet européen SUPERB (https://forest-restoration.eu/).
Résultats et discussion
Au total, 309 toiles d’araignées ont été collectées et un sous-ensemble de 156 échantillons a pu être analysé de manière préliminaire. L’analyse du gène mitochondrial 12S a permis d’obtenir 362 détections de vertébrés non humains réparties dans 108 échantillons positifs. Ces détections incluent 255 occurrences d’oiseaux (Aves), 96 de mammifères (Mammalia), 8 de reptiles (Reptilia) et 3 d’amphibiens (Amphibia), correspondant respectivement à 28, 12, 2 et 2 taxons. En restreignant l’analyse aux seuls vertébrés sauvages, 270 détections ont été recensées dans 78 échantillons.
Parmi les échantillons ayant permis au moins une détection de vertébré sauvage, la richesse taxonomique moyenne par échantillon est plus élevée en forêt de feuillus (moyenne : 4,8 ; variance : 17,4) que dans les haies (2,4 ; variance : 5,8) ou en lisière de pinède (2,0 ; variance : 4,4). Les courbes d’accumulation de taxons semblent confirmer ces différences : la richesse augmente plus rapidement en forêt de feuillus, suivie par les haies puis les pinèdes (Figure 2). Ces résultats sont cohérents avec nos attentes. Les forêts de feuillus, relativement préservées, présentent une richesse élevée à l’inverse des lisières de pin, intensivement exploitées et à végétation peu diversifiée. Les haies semblent occuper une position intermédiaire, contribuant positivement à la biodiversité sans atteindre les niveaux observés en milieux forestiers.
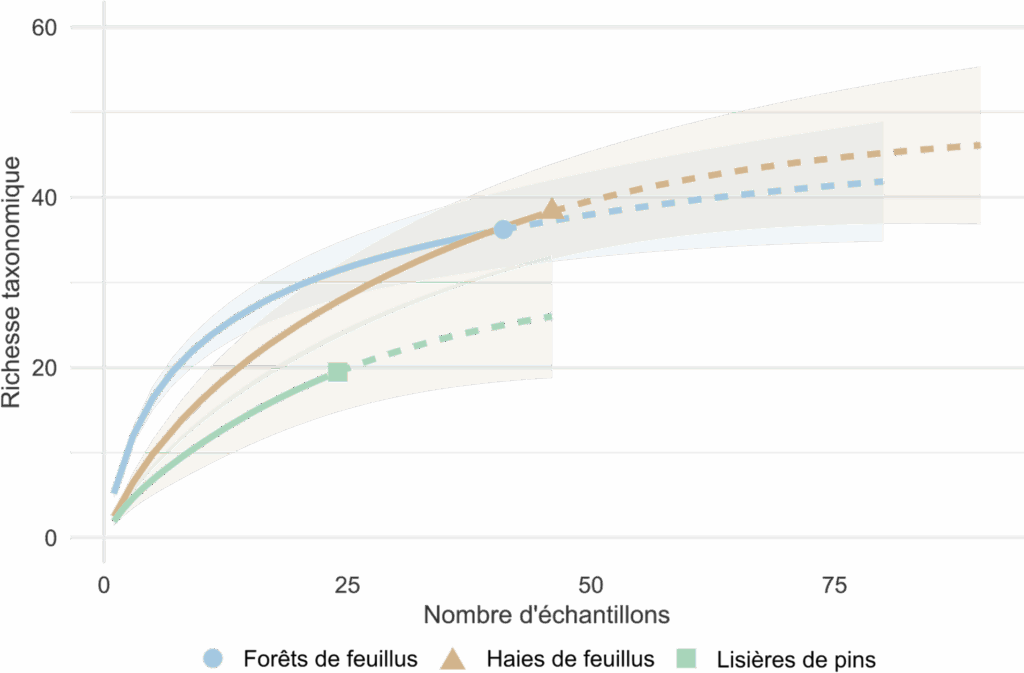
Figure 2 : Richesse taxonomique des vertébrés sauvages selon le type d’habitat Courbes de raréfaction (moyenne ± erreur standard) montrant la richesse en taxons de vertébrés sauvages détectés dans chaque type d’habitat. Les lignes pleines indiquent la richesse observée, tandis que les lignes pointillées correspondent à la richesse extrapolée. Les données sont issues des séquences amplifiées avec le marqueur 12S-V5.
L’amplification du marqueur génétique COI sur le même sous-ensemble a permis d’identifier quatre familles d’araignées (Agelenidae, Araneidae, Linyphiidae et Theridiidae), réparties en neuf genres. Toutefois, seuls 18 % des échantillons contenaient des ASVs affiliés à l’ordre Araneae. Ce taux de détection relativement faible pourrait résulter du manque de spécificité du marqueur utilisé. Sur les 9119 ASVs obtenus, seuls 878 ont pu être assignés taxonomiquement, dont 31 aux Araneae. Le jeu de données est dominé par des ASVs de petits insectes, vraisemblablement issus du plancton aérien, ainsi que par des amplifications non spécifiques incluant des nématodes, mollusques et autres taxons non ciblés.
Ces résultats préliminaires suggèrent que les toiles d’araignées peuvent constituer un substrat utile pour la détection d’ADNe de vertébrés et permettre une caractérisation différenciée de la biodiversité selon les types d’habitats, notamment dans un contexte de restauration écologique. En revanche, l’identification moléculaire des araignées à l’origine des toiles apparaît limitée avec le marqueur utilisé, ce qui restreint pour l’instant l’exploration des relations entre espèces à l’origine des toiles et performances de détection. L’approche typologique, fondée sur la morphologie et la structure des toiles, reste néanmoins prometteuse pour explorer les effets de ces caractéristiques sur la détection d’ADNe.
Dans l’ensemble, ces données renforcent l’idée que les toiles d’araignées peuvent enrichir les dispositifs de biosurveillance terrestre, non seulement pour des comparaisons à l’échelle du paysage, mais potentiellement aussi pour détecter des variations locales de biodiversité. Des analyses plus approfondies sont toutefois nécessaires pour identifier précisément les paramètres influençant la probabilité de détection et pour évaluer la complémentarité de cette approche par rapport aux méthodes traditionnelles.
Références
1. WWF. Living Planet Report – A System in Peril. (2024).
2. Jaureguiberry, P. et al. The direct drivers of recent global anthropogenic biodiversity loss. Sci. Adv. 8, eabm9982 (2022).
3. Thomsen, P. F. & Willerslev, E. Environmental DNA – An emerging tool in conservation for monitoring past and present biodiversity. Biol. Conserv. 183, 4–18 (2015).
4. Nagler, M., Podmirseg, S. M., Ascher-Jenull, J., Sint, D. & Traugott, M. Why eDNA fractions need consideration in biomonitoring. Mol. Ecol. Resour. 22, 2458–2470 (2022).
5. Pawlowski, J., Apothéloz-Perret-Gentil, L. & Altermatt, F. Environmental DNA: What’s behind the term? Clarifying the terminology and recommendations for its future use in biomonitoring. Mol. Ecol. 29, 4258–4264 (2020).
6. Koziol, A. et al. Environmental DNA metabarcoding studies are critically affected by substrate selection. Mol. Ecol. Resour. 19, 366–376 (2019).
7. Collins, R. A. et al. Persistence of environmental DNA in marine systems. Commun. Biol. 1, 185 (2018).
8. Ryan, E., Bateman, P., Fernandes, K., van der Heyde, M. & Nevill, P. eDNA metabarcoding of log hollow sediments and soils highlights the importance of substrate type, frequency of sampling and animal size, for vertebrate species detection. Environ. DNA 4, 940–953 (2022).
9. Lynggaard, C. et al. Airborne environmental DNA for terrestrial vertebrate community monitoring. Curr. Biol. 32, 701-707.e5 (2022).
10. Eberhard, W. Spider Webs: Behavior, Function, and Evolution. (University of Chicago Press, 2020). doi:10.7208/9780226534749.
11. Newton, J. P., Nevill, P., Bateman, P. W., Campbell, M. A. & Allentoft, M. E. Spider webs capture environmental DNA from terrestrial vertebrates. iScience 27, 108904 (2024).
12. Berard, A., Pradel, J., Galan, M. & Charbonnel, N. Spider webs, soil or leaf swabs to detect environmental DNA from terrestrial vertebrates: what is the best substrate? (2025).
13. Newton, J. P. et al. Evaluation of Extraction Methods for the Recovery of Vertebrate DNA From Spider Webs. Environ. DNA 7, e70122 (2025).
14. Escudié, F. et al. FROGS: Find, Rapidly, OTUs with Galaxy Solution. Bioinformatics 34, 1287–1294 (2018).
Utilisation de la bourse
La totalité de la bourse a été utilisée pour l’hébergement et les déplacements des deux personnes chargées de la collecte des toiles d’araignées.


Commentaires récents